
Covid STArS
Covid STArS
Il vantaggio di conoscersi
Il primo test molecolare per Covid-19 basato sul sequenziamento, per l’identificazione completa delle varianti di SARS-CoV-2.
Covid-19 STArS (Strain-Amplicon-Sequencing) è un test per caratterizzare le varianti del virus Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2) mediante sequenziamento, a partire da tamponi e altri campioni clinici oppure da RNA già estratto.
Perchè è importante farlo?

Identificazione delle varianti di SARS-CoV-2
Covid-19 STArS è il primo test commerciale basato sul sequenziamento che permette di identificare in modo rapido e accurato le varianti di virus SARS-CoV-2 responsabili del Covid-19.

È sempre aggiornato
A differenza dei test molecolari e dei test antigenici che devono essere messi a punto per le nuove varianti, Covid-19 STArS può analizzare eventuali nuove regioni del genoma che caratterizzano le varianti del virus senza impatto sui costi e sulle tempistiche di sviluppo.

Non necessita di validazione
I test basati su RT-qPCR e i test antigenici non identificano in modo univoco le varianti di virus SARS-CoV-2 e necessitano quindi di validazione mediante sequenziamento. Covid-19 STArS si basa direttamente sul sequenziamento del genoma virale.
Covid-19 STArS migliora la diagnosi di Covid-19

Perchè?
L’attuale metodo raccomandato per la diagnosi molecolare di Covid-19 si basa sul rilevamento dell’RNA virale in campioni oro-nasofaringei o di espettorato, usando il metodo realtime RT-qPCR. In alternativa possono essere impiegati test antigenici per ottenere diagnosi preliminari in tempi rapidi.
Questi saggi sono sviluppati per riconoscere alcune varianti del virus, ma sono tuttavia incapaci di riconoscere le nuove varianti che sempre più frequentemente si diffondono nella popolazione, un’informazione che si sta dimostrando molto importante in relazione alla diffusione del virus e all’efficacia dei trattamenti (1,2).
Come
A differenza dei test molecolari e antigenici, il test Covid-19 STArS si basa sul sequenziamento, sia per rilevare la presenza dell’RNA virale sia per identificare le varianti genetiche.
Il sequenziamento offre vantaggi consistenti rispetto ai metodi RTqPCR/antigenici, pur mantenendo sensibilità e specificità simili (Figura 1), in particolare:
- è l’unico approccio che permette l’identificazione in modo univoco di tutte le varianti di virus (o ceppi virali) attualmente circolanti.
- Non necessita di validazione perché sequenzia direttamente quelle porzioni del genoma virale che caratterizzano le diverse varianti.
- Può essere rapidamente aggiornato per la rilevazione di nuove varianti.
- Permette di svicolarsi dalla catena di approvvigionamento di reagenti per eseguire test molecolari/antigenici su cui insiste attualmente l’intero pianeta.

Perchè?
L’attuale metodo raccomandato per la diagnosi molecolare di Covid-19 si basa sul rilevamento dell’RNA virale in campioni oro-nasofaringei o di espettorato, usando il metodo realtime RT-qPCR. In alternativa possono essere impiegati test antigenici per ottenere diagnosi preliminari in tempi rapidi.
Questi saggi sono sviluppati per riconoscere alcune varianti del virus, ma sono tuttavia incapaci di riconoscere le nuove varianti che sempre più frequentemente si diffondono nella popolazione, un’informazione che si sta dimostrando molto importante in relazione alla diffusione del virus e all’efficacia dei trattamenti (1,2).
Come
A differenza dei test molecolari e antigenici, il test Covid-19 STArS si basa sul sequenziamento, sia per rilevare la presenza dell’RNA virale sia per identificare le varianti genetiche.
Il sequenziamento offre vantaggi consistenti rispetto ai metodi RTqPCR/antigenici, pur mantenendo sensibilità e specificità simili (Figura 1), in particolare:
- è l’unico approccio che permette l’identificazione in modo univoco di tutte le varianti di virus (o ceppi virali) attualmente circolanti.
- Non necessita di validazione perché sequenzia direttamente quelle porzioni del genoma virale che caratterizzano le diverse varianti.
- Può essere rapidamente aggiornato per la rilevazione di nuove varianti.
- Permette di svicolarsi dalla catena di approvvigionamento di reagenti per eseguire test molecolari/antigenici su cui insiste attualmente l’intero pianeta.

Il test Covid-19 STArS sfrutta il sequenziamento di terza generazione come metodo di analisi delle varianti di SARS-CoV-2.
Il test Covid-19 STArS sfrutta il sequenziamento di terza generazione come metodo di analisi delle varianti di SARS-CoV-2.
Capacità di rilevazione
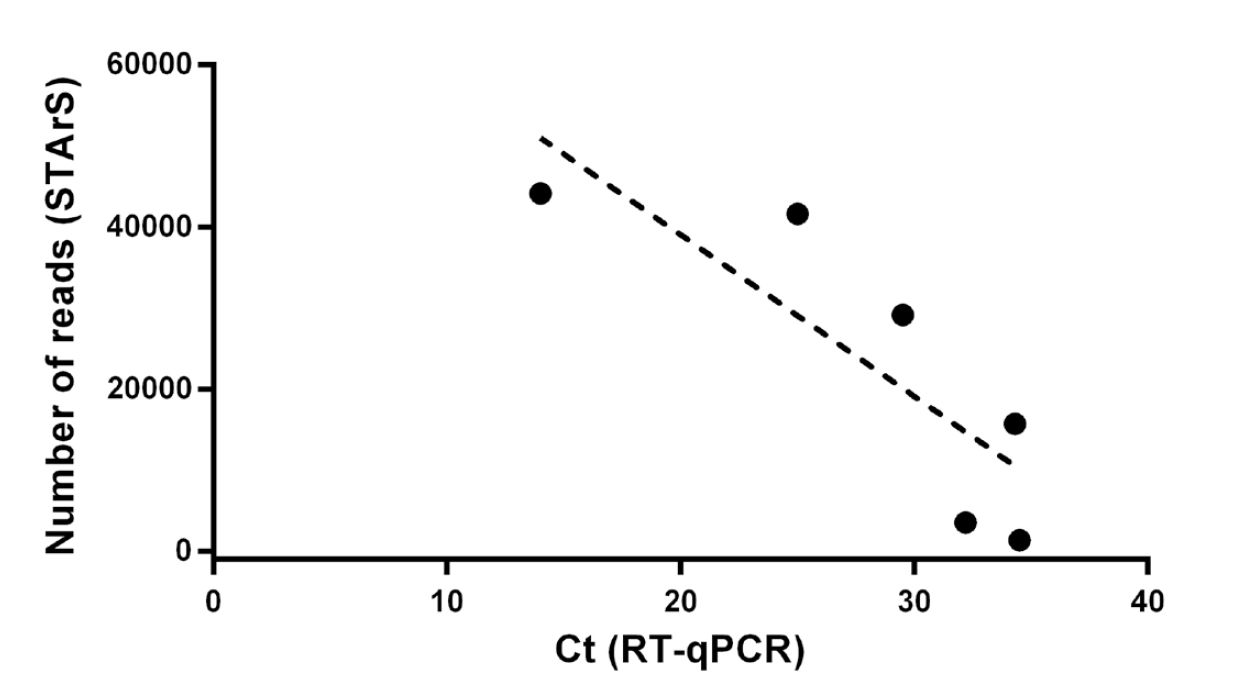
Metodiche a confronto
Covid-19 STArS ha capacità di rilevazione paragonabile a RT-qPCR. Sei campioni clinici di tampone naso-faringeo sono stati analizzati in parallelo mediante RT-qPCR (ThermoScientific) e Covid-19 STArS. Il ciclo soglia di rilevazione ottenuto con RT-qPCR (Ct – RT-qPCR) è stato comparato al numero di reads di sequenziamento prodotte per lo stesso mediante campione Covid-19 STArS (Reads – STArS). L’analisi ha identificato un’ottima concordanza tra le metodiche.
Come funziona?
Covid-19 STArS è un nuovo test diagnostico per SARS-CoV-2 che combina la RT-PCR one-step con il sequenziamento rapido di 10 regioni che caratterizzano i ceppi virali (anche chiamati varianti o sottotipi virali) attualmente circolanti.

Campione di partenza
Il test può essere eseguito a partire da campioni di tamponi oro-nasofaringei, da altri campioni clinici positivi al SARS-CoV-2 o da RNA già estratto.
Amplificazione del genoma virale
In seguito alla retro-trascrizione dell’RNA, Covid-19 STArS amplifica contemporaneamente 10 regioni rilevanti del genoma di SARS-CoV-2:
- 2 regioni corrispondenti a quelle analizzate dai test molecolari tradizionali: geni N e RdP.
- 8 regioni che permettono di identificare le varianti genetiche che caratterizzano i principali ceppi di virus SARS-CoV-2: Inglese, Sud Africana, Brasiliana, Nigeriana, Ugandese.

Sequenziamento
I prodotti di amplificazione vengono analizzati base-per-base mediante sequenziamento rapido impiegando la tecnologia di Oxford Nanopore Technologies.

Analisi dei dati
I dati di sequenziamento vengono analizzati mediante pipeline bioinformatiche messe a punto da Genartis in collaborazione con l’Università degli Studi di Verona, per identificare la presenza del virus e la variante responsabile dell’infezione.

Risultati
Il risultato del sequenziamento sarà reso disponibile entro 72-96h dalla consegna dei campioni (sabato e festivi esclusi), salvo imprevisti.
Un test sempre aggiornato
Dal momento che il virus SARS-CoV-2 è in costante evoluzione, Covid-19 STArS viene continuamente aggiornato sulla base delle informazioni pubblicate nel sito ufficiale del Centro Americano per la Prevenzione ed il Controllo delle malattie (CDC) per permettere la rilevazione di nuove varianti1.
B.1.1.7
Nome comune del ceppo: variante Inglese
Casi in Italia: sì
Nazioni interessate: >90
Mutazioni principali:
Delezione 69/70, Delezione 144Y, N501Y, A570D, D614G, P681H
Caratteristiche riportate:
- Aumento trasmissione3,4,5,6
- Aumento severità di malattia e rischio di ospedalizzazione6,7
- Aumento mortalità (da confermare)6,7,9
- Capacità di alterare test diagnostico10
B.1.351
Nome comune del ceppo: variante Sud-Africana
Casi in Italia: sì
Nazioni interessate: >40
Mutazioni principali:
K417N, E484K, N501Y, D614G
Caratteristiche riportate:
- Diminuzione della suscettibilità agli anticorpi policlonali e monoclonali11
P.1
- Capacità di eludere l’immunità naturale (da confermare)12
- Diminuzione della suscettibilità agli anticorpi policlonali e monoclonali11
A.23.1
Nome comune del ceppo: variante Ugandese
Casi in Italia: /
Nazioni interessate: >20
Mutazioni principali:
F157L, V367F, Q613H, P681R
Caratteristiche riportate: /
B.1.525
Nome comune del ceppo: variante Nigeriana
Casi in Italia: sì
Nazioni interessate: >30
Mutazioni principali:
E484K, Q677H
Caratteristiche riportate: /

B.1.617
Nome comune del ceppo: variante Indiana
Casi in Italia: sì
Nazioni interessate: ~25
Mutazioni principali:
D111D, G142D, L452R, E484Q, D614G e P681R
Caratteristiche riportate:
- Ipotizzato aumento trasmissione (da confermare)13

Perchè scegliere Genartis
l team di Genartis è costituito da un gruppo di genetisti che collabora da molti anni con numerosi ospedali italiani per condurre analisi genomiche su pazienti e loro famiglie. Il team è inoltre coinvolto in progetti di ricerca nazionali ed internazionali (es. 1+M Genomes initiative) per garantire l’identificazione e l’implementazione delle pratiche analitiche più aggiornate per questo tipo di analisi.
Genartis offre la possibilità di avvalersi di una consulenza con un medico genetista prima e dopo l’esecuzione dei test, questo garantisce un’analisi personalizzata del genoma e l’identificazione dei rischi legati alla propria salute.
ExomeTest e GenomeTest richiedono un semplice prelievo di saliva e forniscono risultati paragonabili a quelli ottenuti tramite prelievo venoso.
Contattaci


